Entzündung ist ein Markenzeichen von vielen gesundheitlichen Bedingungen, aber die Quantifizierung, wie die zugrunde liegende Biologie der Entzündung trägt zu bestimmten Krankheiten hat, war schwierig. Für die erste Zeit, UNC School of Medicine Forscher und Kollegen berichten nun über die Entwicklung einer neuen Technologie zum identifizieren von weißen Blutkörperchen, den sogenannten Neutrophilen Granulozyten, die sind grundiert Auswerfen entzündliche DNA in die Zirkulation über einen Prozess namens NETosis.
Die Ergebnisse, veröffentlicht in Wissenschaftliche Berichte, zum ersten mal haben Wissenschaftler verwendeten machine learning-tools für die schnelle, qualitative und quantitative zellanalyse in der Grundlagenforschung.
„Dieser neue test wird erlauben, die Ermittler zu Messen NETosis in verschiedenen Krankheiten und zum test von Medikamenten, hemmen oder fördern den Prozess,“ sagte senior-Autor Leslie Parise, Ph. D., professor und Stuhl der UNC-Abteilung von Biochemie und Biophysik.
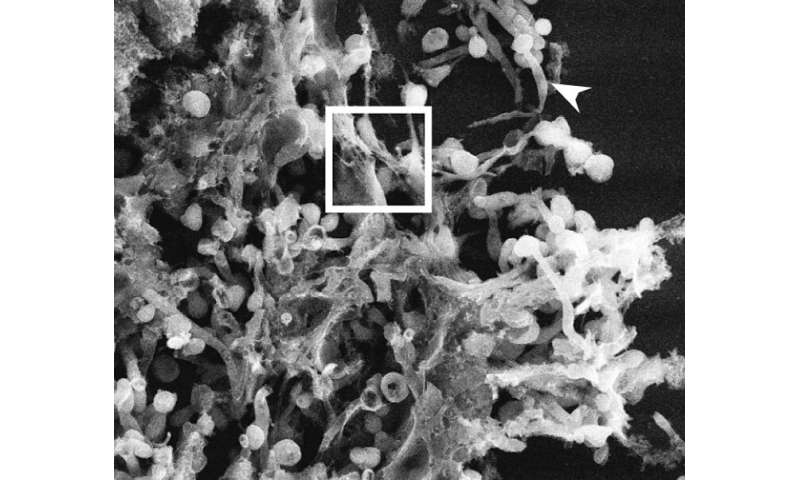
Wenn fremde Eindringlinge wie Viren oder Bakterien geben Sie unserem Körper die weißen Blutkörperchen eilen im Kampf gegen die Invasoren auf verschiedene Weise. Ein Typ weißer Blutkörperchen, der Neutrophilen, vertreibt seine DNA in die Blutbahn trap Bakterien und Viren und helfen, Ihre Tötung zu verhindern, dass Infektionen. Diese Neutrophilen DNA hat eine netzartige Aussehen und wird als Neutrophile Extrazelluläre Fallen oder NETs. Der Prozess, durch den diese DNA-Schub in den extrazellulären Raum als NETosis. Diese sogenannte DNA-Netze sind gemeldet, dazu beitragen, Entzündungen in zahlreichen Krankheiten, wie Autoimmun-Krankheit, sepsis, arthritis, Krebs, Sichelzellanämie und Thrombose.
NETosis kann aktiviert werden durch verschiedene Chemische Reize, aber für das Auge, die Letzte NETotic Neutrophilen gleich aussieht. Zu helfen, zu unterscheiden Neutrophile Granulozyten, aktiviert durch verschiedene stimuli und in eine sehr schnelle Art und Weise, die Labor Parise lehnte sich auf der Maschine lernen, ein Zweig der künstlichen Intelligenz basiert auf der Idee, dass Computer erwerben wissen durch Daten und Beobachtungen ohne explizite Programmierung. Die Computer können dann lernen, das verallgemeinern von Beispielen und Vorhersagen zu machen.
Maschinelles lernen verwendet wurde, in der Analyse, genomics, drug discovery, Modellierung von protein-Strukturen und der Diagnose von Krankheiten. Nur wenige Studien haben verwendet, automatisierte imaging-Technologien, machine learning in non-diagnostische und explorative Forschung-konzentrierte Bemühungen, wie Zell-Quantifizierung in Tier-Modellen.
„Die machine-learning-revolution hat kommt erst in den letzten Jahren aufgrund der rasanten Entwicklungen in parallel computing and mathematical optimization-theory“, sagte co-Autor Joshua Cooper, Ph. D., Professor in der Abteilung von Mathematik an der Universität von South Carolina. „Die meisten wissenschaftlichen arbeiten über diese außerordentlich leistungsfähige Werkzeuge, so weit hat sich auf den Aufbau von Vorhersagemodelle, und in der Regel auf sehr teure Ausrüstung. Wir haben gezeigt, es ist auch möglich, um die Bereitstellung dieser tools, die auf commodity-hardware Voraus grundlegenden Wissenschaft durch die Automatisierung von Analysen, die zuvor erfordern enorme Mengen an menschlicher Arbeitskraft, und durch die Umwandlung der qualitativen biologischen Morphologie in messbar quantitative Merkmale.“
Zelle Klassifizierung ist mühsam, sich auf stark überwacht-Bild-Analyse-tools mit der Notwendigkeit einer kontinuierlichen Interaktion des Benutzers. Obwohl das reduziert die Fehlerquote und außergewöhnlichen Lern-Geschwindigkeiten des maschinellen Lernens, haben das Potenzial zu verändern, auf dem Gebiet der zellulären Bildgebung, haben Sie nicht weit verbreitet in den biologischen Wissenschaften, in Teil, weil der Mangel an Prüfung und Validierung solcher Methoden aufgrund der Verknappung von großen Datenmengen für die Ausbildung.
Convolutional neural networks (CNNs) sind deep-learning-algorithmen Häufig in der Bilderkennung und Klassifizierung. Ihre Struktur wurde inspiriert durch die Struktur des mammalian visual cortex, deren Funktion es ist, Mustererkennung und computing-komplexe Objekt-Attribute.
In der Wissenschaftlichen Berichte, paper, ersten Autor Laila Elsherif, Ph. D. und Kollegen, einschließlich der co-Autoren Noah Sciaky bei UNC und Joshua Cooper, Ph. D., der Universität von South Carolina, zeigte zum ersten mal die Möglichkeit der Gestaltung von verschiedenen CNNs für die Lösung wichtiger Fragen der Neutrophilen NETosis.
Elsherif, wer ist jetzt ein Mitglied der Fakultät an der Universität von Tennessee, auf diese neue Technologie, um die sichelzellkrankheit (SCD), da chronische Entzündung und hypercoagulability sind bekannte Komplikationen bei SCD-Patienten, und die Netze werden als wichtig für beide, Entzündung und Blutgerinnung. Auch haben frühere Studien festgestellt, dass plasma von SCD-Patienten verursacht, der NET-Produktion in Neutrophilen von gesunden Personen führt zu dem Schluss, dass NETosis ist im Zusammenhang mit SCD Pathophysiologie.
Aber beide Studien verwendeten Surrogat-Indikatoren für NETosis und nicht Messen, NETosis direkt in Neutrophilen Granulozyten isoliert von SCD-Patienten.
„Unsere Technologie ermöglicht es uns, quantitativ zu bewerten NETosis in Neutrophilen Granulozyten von Patienten mit SCD, in denen die Patienten nicht erleben Schmerzen und andere Symptome im Zusammenhang mit Krise,“ Elsherif sagte.
Die Ermittler angewendet, Ihre neue Technologie zu finden, die einem Pfad der NETosis scheint abwesend zu sein, in der Patienten getestet.
